- Programmbereich 4
Immun-Epigenetik

- Epigenetische Prägung
- Epigenetische Regulation
- T-Lymphozyten
- Zelltherapie
Das Epigenom in T-Zellen verstehen und therapeutisch nutzbar machen
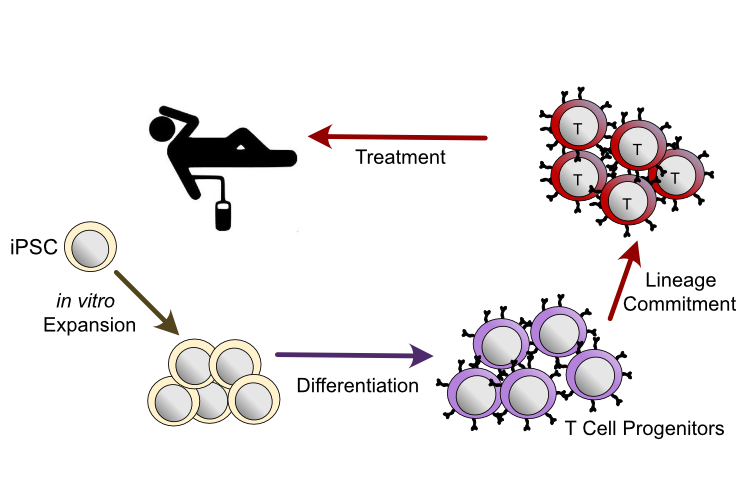
Für viele rheumatische Erkrankungen existieren bisher keine heilenden Therapien, wodurch die Patienten auf lebenslange immunsuppressive Behandlungen angewiesen sind. Adoptive Zelltherapien bieten aber eine vielversprechende Strategie um Heilung – also die vollständige Wiederherstellung der immunologischen Homöostase – zu erreichen. Dabei gibt es zwei aussichtsreiche Strategien: einerseits eine dauerhafte Stärkung des immunsuppressiven Arms des Immunsystems (durch sogenannte Treg-Therapie) und andererseits die Eliminierung des pathogenen autoreaktiven Teils (z.B. mit CAR-T-Zell-Therapie).
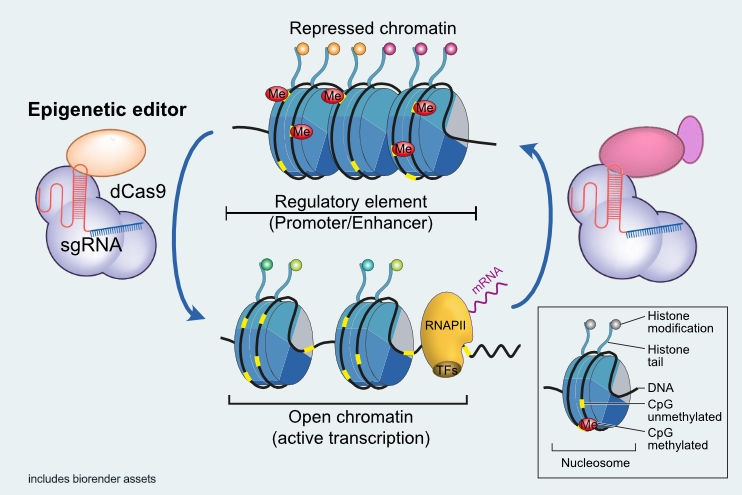
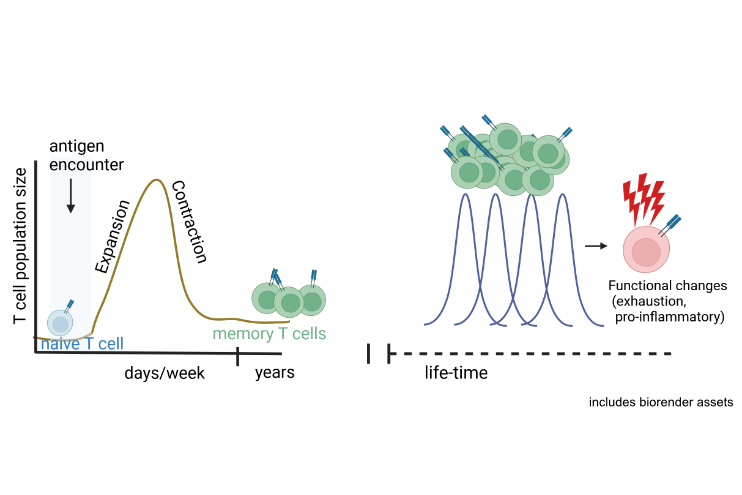
Unsere Arbeitsgruppe untersucht die epigenetischen Mechanismen, die die Differenzierung, Funktion und Seneszenz von T-Zellen bei Gesundheit und Krankheit steuern. Aus den gewonnenen Erkenntnissen wollen wir Ansätze für gezielte epigenetische Interventionen entwickeln, um neue Zelltherapien für Autoimmun- und chronisch-entzündliche Erkrankungen zu schaffen. Dabei nutzen wir das regulatorische Potenzial, das durch die dreidimensionale Faltung des Genoms vermittelt wird, um T Zellen in optimale Funktionszustände für Zelltherapien zu versetzen.
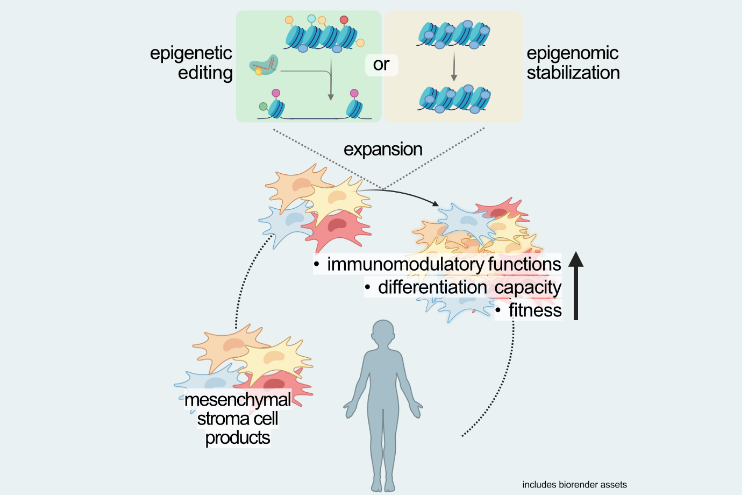
Zu diesem Zweck nutzt unsere Gruppe ein breites Portfolio an Techniken zur epigenetischen Charakterisierung sowohl ganzer Genome als auch spezifischer Zielgene, die auch für klinischen Proben anwendbar sind. Darüber hinaus entwickelt und wendet unsere Gruppe innovative Ansätze zur gezielten Modifikation epigenomischer Strukturen an, um therapeutische Zellprodukte zu optimieren.
Durch die Verbindung von epigenetischer Grundlagenforschung mit angewandten therapeutischen Strategien will unsere Gruppe epigenetisch-optimierte Zelltherapien für immunvermittelte Krankheiten entwickeln.